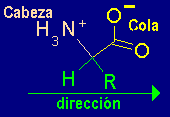
Estructura y propiedades del enlace peptídico.
Un péptido es un polimero de aminoácidos. Un polimero (de poli, muchos y meros, unidad) es una cadena de unidades básicas unidas entre sí por enlaces químicos covalentes.
(ver un dibujo animado de la formación de un polímero)
Hay muchos tipos de polímeros y un buen ejemplo de un polímero simple es el polietileno. En el polietileno, la unidad básica es el etileno CH2=CH2. Así, por ejemplo, un dietiléno estaría formado por 4 átomos de carbono H3C-CH2-CH2-CH3 y sería equivalente al gas butano. Estos polímeros pueden tener cadenas realmente largas, como es el caso del polietileno que usamos para las bolsas de plástico.
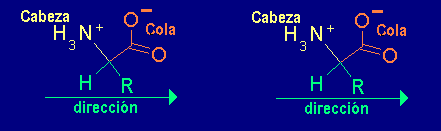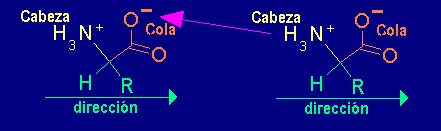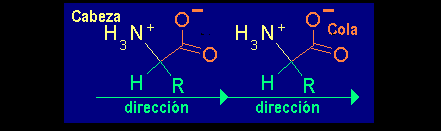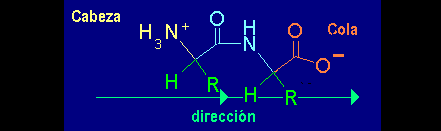
A diferencia del etileno, los L-a-aminoácidos poseen en común dos grupos funcionales distintos y por ello es posible designar a uno de ellos (el grupo a-amino) como la cabeza y al otro (el carboxilato) como la cola:

Hay varias maneras de unir a dos aminoácidos entre sí, pero si queremos tener siempre un nuevo sitio de unión para añadir un tercer aminoácido debemos unirlos cabeza con cola, dejando así la cabeza del primero y la cola del segundo libres para reaccionar con nuevas unidades de aminoácido:
Una cadena de hidrocarburo lineal como el polietileno es perfectamente simétrica y no se puede distinguir el principio del final. un polímero de aminoácidos tiene un inicio y un fin. Por ello, en un polímero simétrico, no es posible codificar información, porque no sabríamos en que lado empezar a leer.
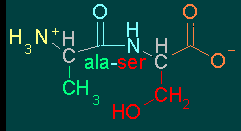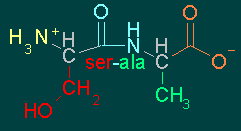
Sin embargo, en los péptidos el dipéptido ala-ser es diferente del ser-ala, ya que el primero resulta de unir el grupo -COOH de la alanina (ala, A) con el -NH2 de la serina (ser, S), mientras que el segundo resulta de unir el grupo -COOH de la serina con el -NH2 de la alanina.
Al reaccionar un ácido carboxílco con una amina, el enlace resultante se denomina una amida y es semejante al grupo amídico polar que encontramos en la cadena lateral de los aminoácidos asparagina (asn, N) y glutamina (gln, Q). El enlace amida formado entre dos a-aminoácidos recibe el nombre de enlace peptídico. Los aminoácidos unidos por enlaces peptídicos ya no pueden llamarse aminoácidos.
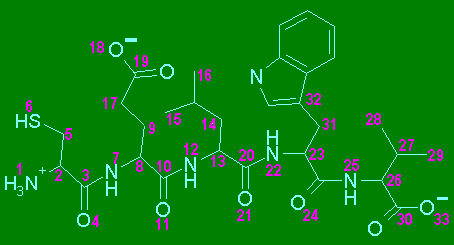
La cadena formada por varios aminoácidos unidos recibe el nombre de polipéptido y para referirnos al conjunto de átomos formado por un nitrógeno imidico, Ca, un carbonilo y su grupo R decimos que se trata de un resíduo de aminoácido.
Identifique los átomos que conforman el tercer resto de aminoácido en el siguiente pentapéptido:
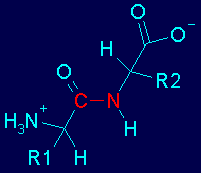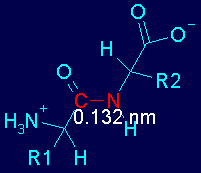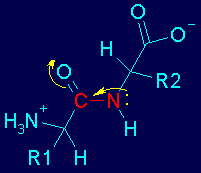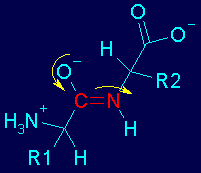
Las amidas poseen varias propiedades singulares, que derivan del hecho de que el orbital que alberga al par de electrones no compartidos del átomo de nitrógeno se sobrepone con el orbital de tipo p del doble enlace >C=O.
Así, una amida:
Tiene serias dificultades para aceptar un protón, ya que el par de electrones no compartidos no está disponible, es decir, no actúa como base en un medio acuoso (por ello la asparagina y la glutamina no son aminoácidos básicos).
Al poseer el N un par de electrones no compartidos se genran formas resonantes, por lo tanto, el enlace posee cierto caracter de doble enlace, es decir, alta densidad electrónica y un orbital tipo-p.
Esto tiene consecuencias en la geometría de los átomos alrrededor del enlace.
Propiedaes químicas.
Los péptidos al formarse pierden un grupo amino y un carboxilato por cada enlace que se forma, por lo tanto, sólo el primer grupo amino y el último grupo carboxilato, que no han reaccionado pueden ionizarse. Sin embargo, muchos grupos R poseen funciones químicas ionizables que pueden participar en las propiedades ácido base de un péptido.
Por tanto, todos los péptidos poseen tantos pKa's como aminoácidos con grupos R ionizables tengan y además, un amino y un carboxilo.
¿Cuantos valores de pKa posee el pentapéptido F-A-V-G-H-P? (ver la tabla de grupos R ionizables)
La diferencia de electronegatividad entre el átomo de nitrógeno y el hidrógeno unido a este en el enlace peptídico hacen de esta parte del enlace un grupo con capacidad para donar un puente de hidrógeno. Además, el oxígeno del carbonílo puede aceptar hidrógenos en la formación de puentes de hidrógeno.
Así, un enlace peptídico puede aceptar un puente de hidrógeno y donar otro simultánemente. Las restricciones geométicas hacen que esto no pueda ocurrir internamente. Es decir, para que se pueda dar la formación de puentes de hidrógeno se requiere que otros átomos (de la misma o de otra molécula) se acerquen al enlace peptídico.
Gracias a que conservan cierta polaridad los péptidos poseen solubilidad en agua, a pesar de que la formación de la amida elimna los grupos cargados (-COO- y N+H3) y reduce por tanto la polaridad. Sin embargo, dicha solubilidad es marginal, ya que su estructura electrónica de dobles enlaces resonantes, aunada a la presencia de los carbonos a en las esquinas del plano son regiones bastante apolares.
Consecuentemente, los enlaces peptídicos en solución pueden presentar un caracter polar o apolar y el balance dependerá en gran medida de la naturaleza de los substituyentes laterales de los aminoácidos que lo forman. Así. el dipéptido leu-val será insoluble en agua, mientras que el dipéptido glu-lys será bastante soluble. El dipéptido gly-gly posee una solubilida limitada, pero si se disuelve en agua.
Seleccione el dipéptido más soluble de entre los siguientes:
Desde el punto de vista químico, el enlace peptídico es bastante estable y no se hidroliza con facilidad. Para promover la reacción de hidrólisis se requiere un medio ácido ( HCL 6N por ejemplo) y alta temperatura (90oC) por tiempos prolongados (24-72 h). Estas condiciones son tan drásticas que destruyen algunos aminoácidos cuyos grupos R son sensibles, por ejemplo, los aminoácidos asn y gln poseen un grupo R que es también una amida! y el anillo indólico del triptofano también resulta particularmente delicado en estas condiciones.
El método de hidrólisis arriba mencionado ha sido empleado para determinar la composición de aminoácidos que presenta un péptido. Esto quiere decir, determinar cuantos residuos de cada tipo de aminoácido se requieren para formar la secuencia de residuos que presenta un determinado péptido. Cabe aclarar que esto no significa que con esta información podamos determinar la secuencia de aminoácidos de dicho péptido. En estos casos, el contenido de asn y gln es desconocido y sólo podemos determinar cunato glu+gln (glx) y cuanto asp+asn (asx) se encuentra.
La solución de aminoácidos resultante de la hidrólisis de un péptido puede analizarse mediante cromatografía de intercambio iónico o cromatografía en fase reversa.
Una propiedad muy importante de los enlaces peptídicos, es que el nitrógeno amídico puede ser desplazado del enlace peptídico por su reacción con el grupo tiocarbamida en medio ácido. Como una tiocarbamida-N substituida se puede generar a partir de la reacción entre una amina y un isotiocianato (reacción que ocurre en medio básico), es posible hacer reaccionar el extremo amino de un péptido con fenilisotiocianato en medio básico, para formar el péptido feniltiocarbamilado correspondiente. Luego de lavar el exceso de fenilisotiocianato, se aplica un medio ácido y el tiocarbamoilo correspondiente formará un ciclo de 5 miembros llamado feniltiohidantoina con el carbonilo del enlace peptídico adjacente. El resultado es un péptido más corto, con un nuevo extremo amino y la feniltiohidantoina del primer aminoácido. Esta reacción se conoce como degradación de Edman. Puesto que el análisis de la feniltiohidantoina revelará la naturaleza del aminoácido que se encontraba en el extremo amino del péptido original y el nuevo péptido puede someterse al mismo procedimiento para determinar el aminoácido de la siguiente posición, la degradación de Edman es la báse de las técnicas que permiten determinar la secuencia de péptidos
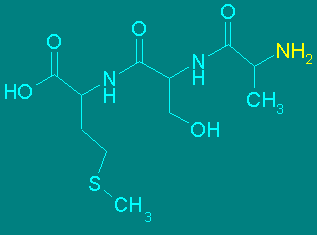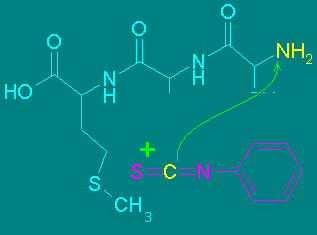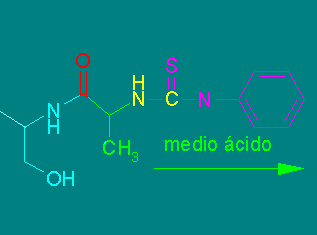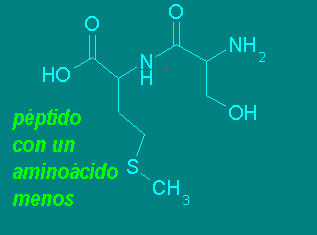
En la práctica la degradación de Edman puede llevarse hasta unos 20 ciclos con cierta facilidad, pero la determinación de secuencias más largas se hace cada vez más difícil. Por ello, para determinar la secuencia completa de los aminoácidos de una proteína por medio de la degradación de Edman, es necesario fragmentar primero la proteína en secciones más pequeñas y luego separar los fragmentos.
| (Índice) | (créditos) | (Aminoácidos) | (Geometría) |